
微生物+单细胞分选仪 拉曼单细胞分选仪 菌落原位多表型检测与挑取工作站 单细胞光镊操纵与分选系统 |
拉曼+共聚焦拉曼光谱仪 近红外共聚焦拉曼光谱仪 拉曼单细胞分选仪 |
成像+超快三维荧光成像系统 智能细胞荧光计数仪制药颗粒物检测仪 |
核酸快检便携式核酸恒温扩增分析仪耗材拉曼信号增强芯片 细胞分选芯片 |

微生物+单细胞分选仪 拉曼单细胞分选仪 菌落原位多表型检测与挑取工作站 单细胞光镊操纵与分选系统 |
拉曼+共聚焦拉曼光谱仪 近红外共聚焦拉曼光谱仪 拉曼单细胞分选仪 |
成像+超快三维荧光成像系统 智能细胞荧光计数仪制药颗粒物检测仪 |
核酸快检便携式核酸恒温扩增分析仪耗材拉曼信号增强芯片 细胞分选芯片 |
2023年1月26日,吉林大学第一医院儿科呼吸科张影医生与中科院长春光机所李备研究员团队应用长光辰英核心产品—P300共聚焦拉曼光谱仪在《Frontiers in Microbiology》杂志上联合发表了题为“Rapid identification and drug resistance screening of respiratory pathogens based on single-cell Raman spectroscopy”的论文,为呼吸道病原菌的检测及耐药菌的快速筛选提供了新的思路。

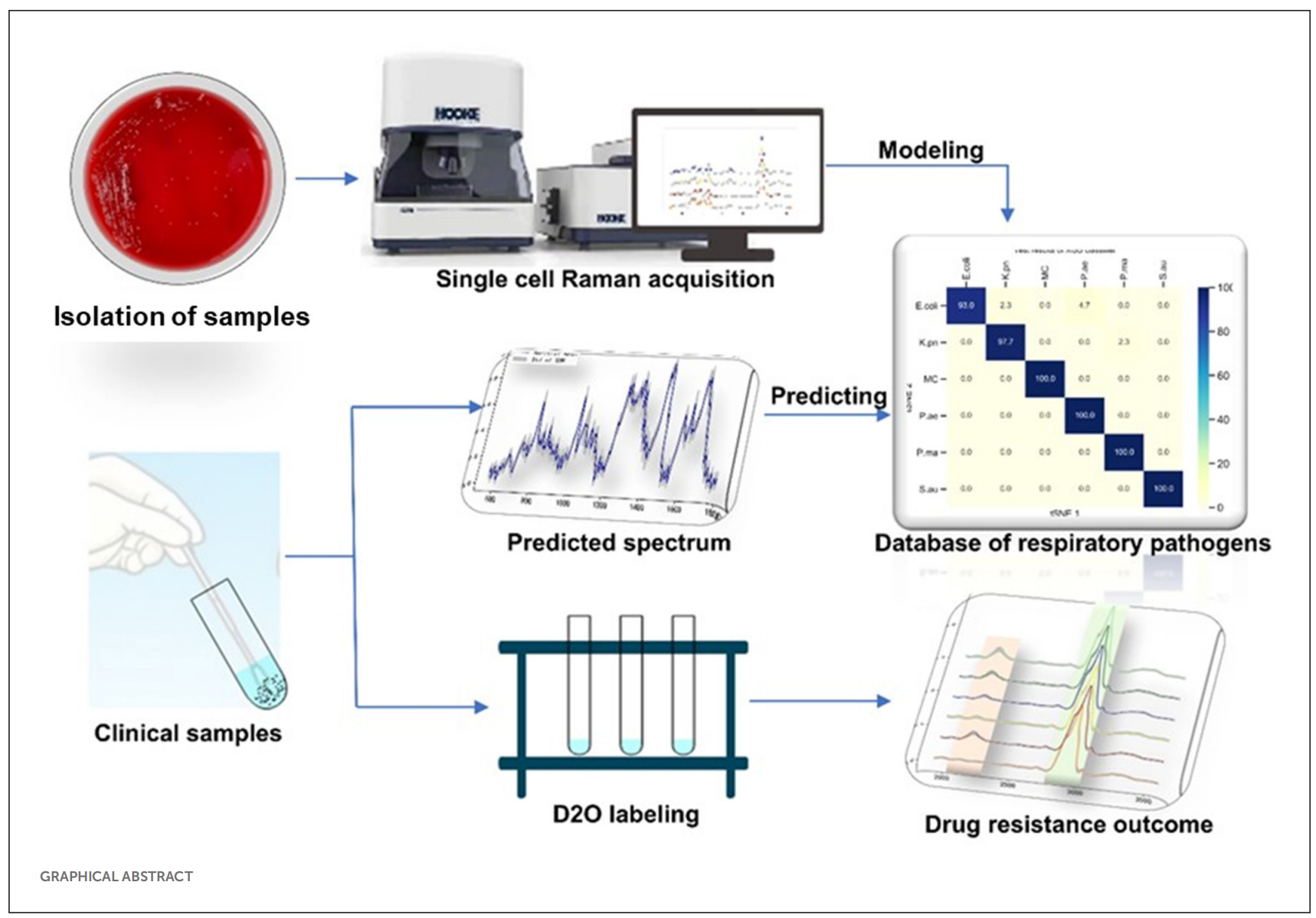
呼吸道感染在全球疾病的经济负担中排名第四,下呼吸道感染是低收入国家的主要死亡原因。针对侵染呼吸道的病原菌进行快速鉴定及耐药性筛查,有助于及时治疗和抗生素的精准选择,因此可以降低下呼吸道感染患者的死亡率。单细胞拉曼光谱是一种“全生物指纹”技术,可用于识别微生物样本。它具有无标记、快速、无损检测的优点。本研究采用单细胞拉曼光谱法收集了6株呼吸道病原体分离株的光谱数据,采用t-SNE分析算法比较了六种呼吸道病原体的差异,采用XGBoost算法建立了拉曼表型数据库模型,并且针对建立的呼吸道病原菌的拉曼表示数据模型进行验证,其分离样本的分类准确率为93-100%,临床样本的分类准确率在80%以上。结合重水标记技术,测定了呼吸道病原体的耐药性。研究表明, SCRS-D?O标记可以在2h内快速识别呼吸道病原体的耐药性。
常见的感染呼吸道病原菌的单细胞拉曼光谱分析:利用单细胞拉曼光谱在单细胞水平上获得微生物拉曼光谱,可以反映病原体的“指纹”特征。为了分析常见呼吸道病原体的生理特征,本研究从临床样本中分离、纯化和培养了6种呼吸道病原菌(E. coli、K. pn、S. au、MC、P. ma、P. ae)。为了建立全面可靠的训练数据集,采集6株菌株的具有代表性的拉曼光谱,并通过数据处理及分析得到的各病原体的特征光谱如图1所示。拉曼光谱指纹图谱(400-1800cm-?)代表了细菌细胞的生化信息,包括核酸、蛋白质和脂质。每种细菌的拉曼光谱都包含了研究细胞复杂结构的必要信息。因此我们针对核酸、蛋白质、脂质这三种成分对应光谱进行深入分析,评估其每种病原菌特定的成分差异。
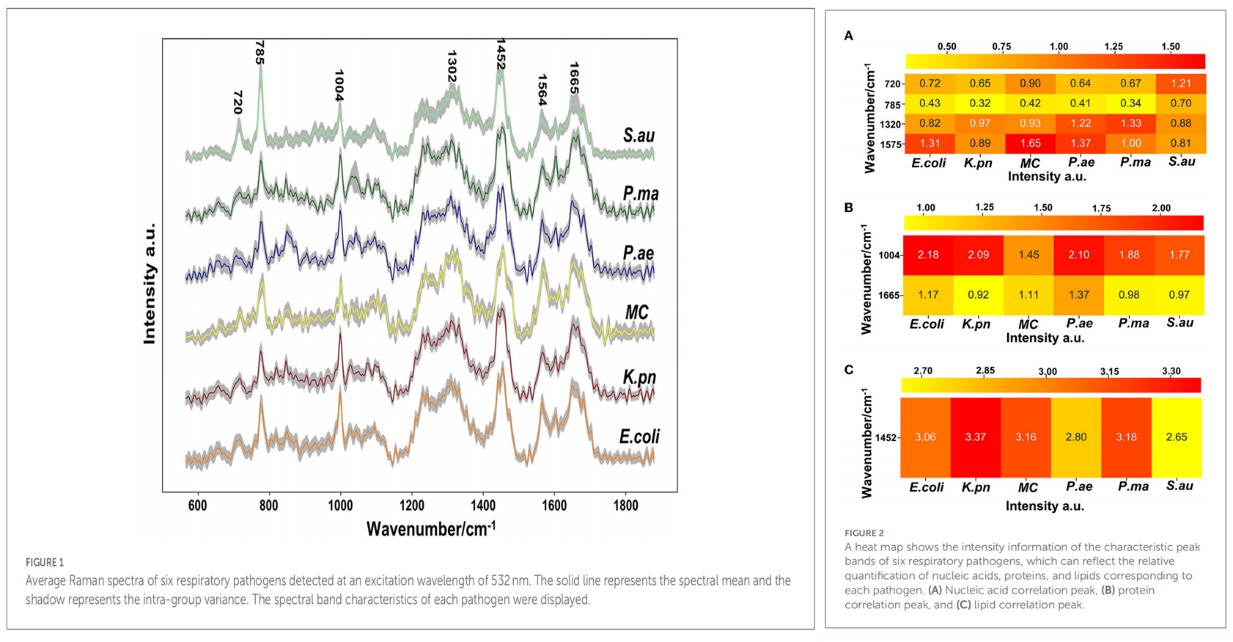
图1. 6种常见呼吸道感染病原菌拉曼光谱特征峰谱及对应特征物质成分差异分析
呼吸道病原菌的差异分析及建立数据模型:为了深入分析6种呼吸道病原菌的之间是否存在显著差异,本文应用t-SNE算法对其数据集进行聚类分析,对高维拉曼光谱降维聚类后从图2的结果上可以得出,同簇类别具有高内聚性,不同簇类别间具有低耦合性。降维后重建t- SNE的前两个特征维度,展示了相关拉曼波段对t-SNE的t-SNE1和t-SNE2贡献的拉曼峰谱。
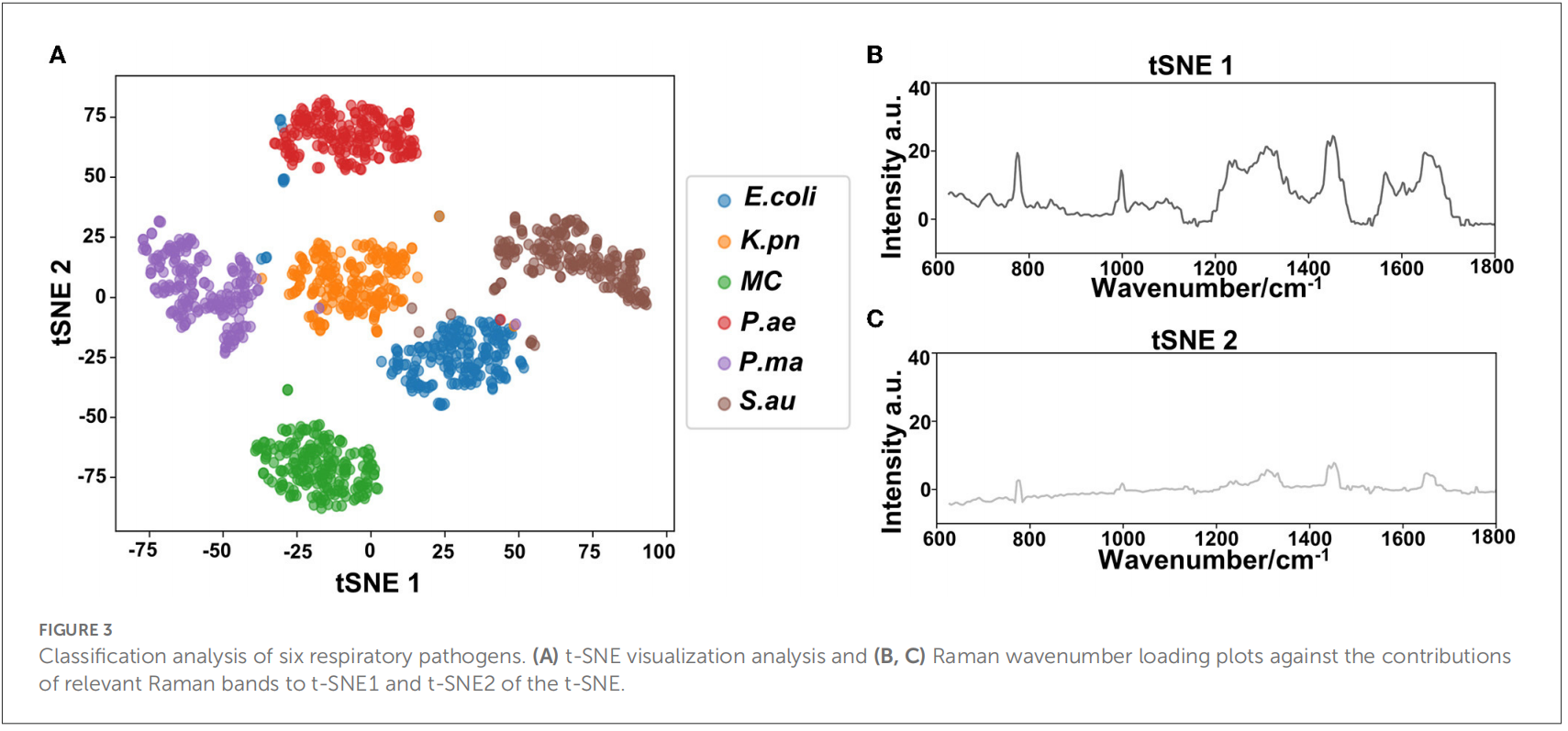
图2. t-SNE算法分析病原菌之间的显著差异
XGBoost是一种经典的综合提升算法框架,具有训练效率高、预测效果好、参数可控、易于使用等特点;它是广泛数据分析领域的尖锐工具,广泛应用于生物医学、环境检测等领域,取得了良好的效果。XGBoost用于揭示拉曼光谱中的复杂信息,并在生物学研究中对细菌进行识别和分类。在本研究中,我们对分类模型进行了训练,建立了6种呼吸道病原菌的拉曼数据模型,并使用再采集的数据对模型进行预测,如图3的结果显示XGBoost对6种呼吸道病原体具有非常准确的预测效果,总体预测准确率为93-100%。
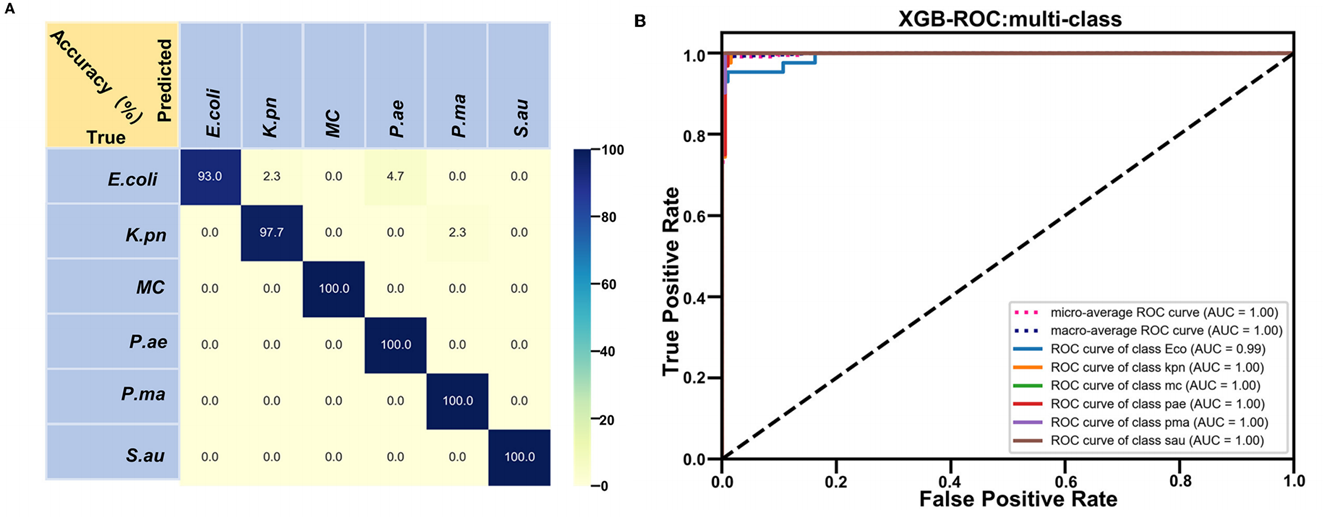
图3. XGBoost算法建立6种呼吸道病原菌拉曼数据模型
拉曼结合重水标记法检测铜绿假单胞菌的耐药性:从6种呼吸道病原菌中选取铜绿假单胞菌进行耐药性的检测,使用重水孵育培养结合拉曼技术可以实现铜绿假单胞菌的耐药性快速筛查。首先对铜绿假单胞菌进行耐药性检测,结果显示与抑菌圈实验一致,如图4所示。并且通过重水标记与拉曼结合方法可以在2h内实现病原菌耐药性筛查。
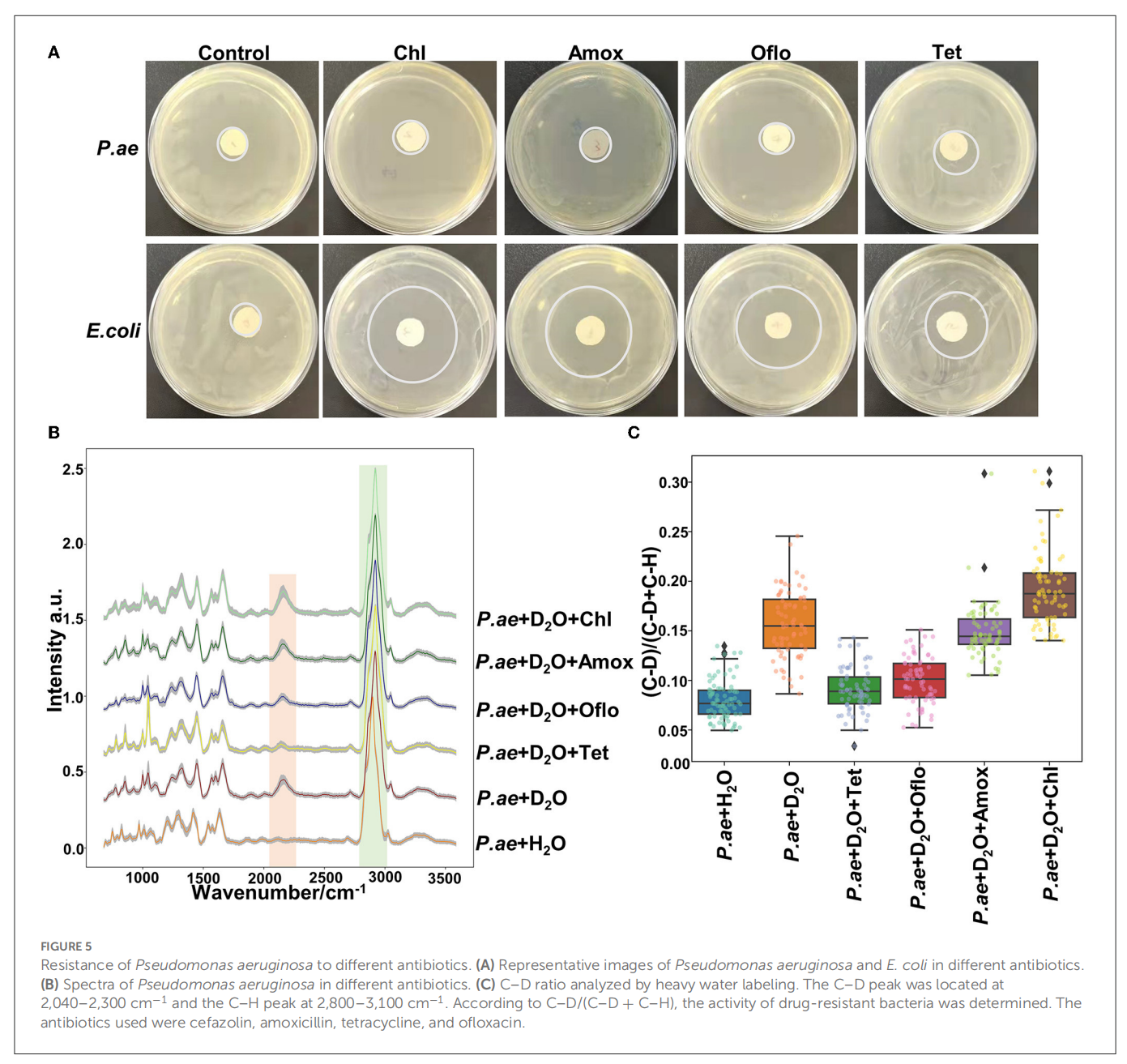
图4.拉曼-重水结合对铜绿假单胞菌抗生素敏感性检测
临床呼吸道感染患者样品鉴定。我们从临床感染患者中收集了6份样本并分培获得其纯菌株,临床鉴定为2株E.coli,2株S.au、2株P.ae。从每个样品中随机抽取10个单细胞进行拉曼光谱检测采用上述建立的XGBoost模型来预测临床分离的样本,该模型对大肠杆菌的预测准确率为80%,对铜绿假单胞菌为90%,对金黄色葡萄球菌为100%。如图5所示。
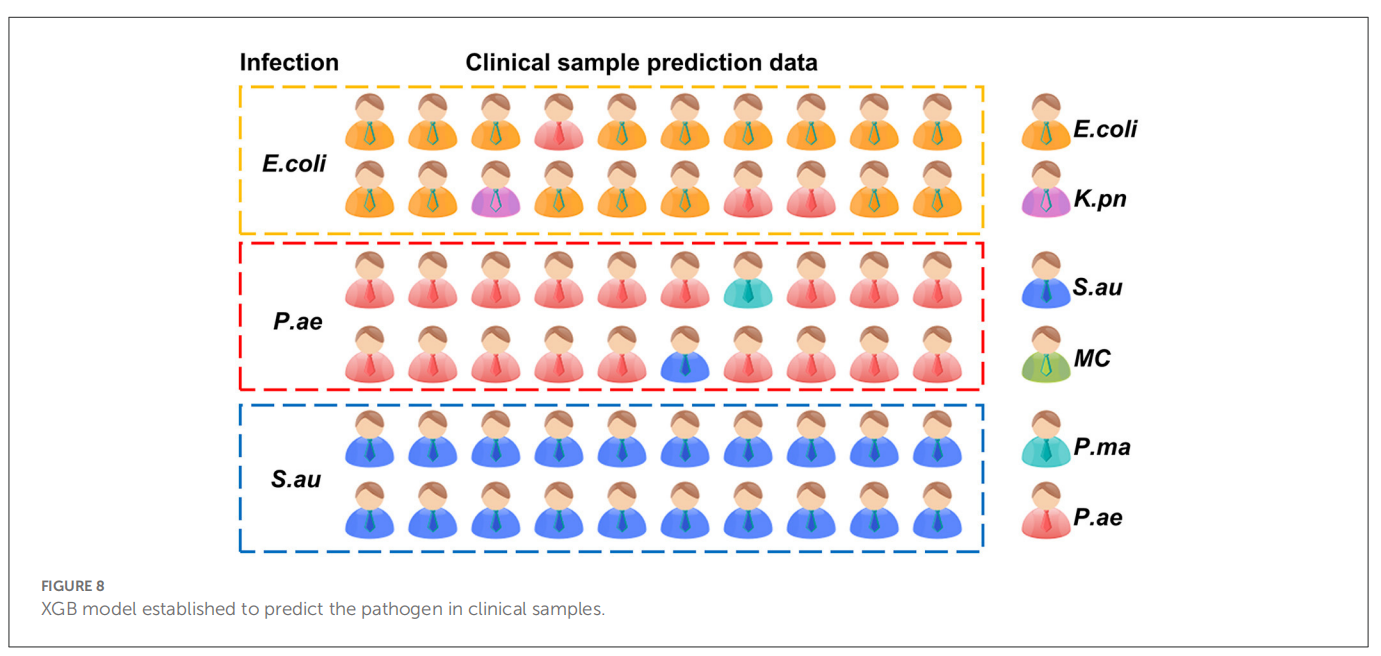
图5. XGBoost模型预测临床样品分离株
本研究采用单细胞拉曼光谱技术对呼吸道病原菌进行检测,应用t-SNE分析算法,分析6种呼吸道病原菌之间的显著性差异,采用XGBoost机器学习分类模型对6种呼吸道病原体进行分类,准确率为93-100%,并且临床呼吸道感染患者的分类准确率在80%以上。拉曼技术结合重水标记可以快速筛查呼吸道病原菌的耐药性,耐药菌的抗性筛查时间可以控制在2h之内。因此,本研究为SCRS-D?O对呼吸道病原体的鉴定和耐药筛查提供了一种新的可行方案。
文献链接:
https://www.frontiersin.org/articles/10.3389/fmicb.2023.1065173/full


 0431-81077008
0431-81077008